conda的安装与使用(2020-08-10更新)
https://bioconda.github.io/recipes ,告诉她安装conda吧,只要一行命令conda install gatk就行了。(2019-04-29 update:这个网址已经挂掉了,请改用:https://anaconda.org/bioconda/repo,或者直接用bing/google搜“conda gatk” 这样的关键词,基本上跳出来的第一个就是。然后她很可爱的问我conda怎么装。。果断就把这篇教程丢给她了,但是发现现在的用法和以前的不是很一样了,所以就把原来的教程更新了一下以飨读者。
conda简介
Conda 是一个开源的软件包管理系统和环境管理系统,用于安装多个版本的软件包及其依赖关系,并在它们之间轻松切换。 Conda 是为 Python 程序创建的,适用于 Linux,OS X 和Windows,也可以打包和分发其他软件。
安装conda
conda分为anaconda和miniconda。anaconda是包含一些常用包的版本(这里的常用不代表你常用 微笑.jpg),miniconda则是精简版,需要啥装啥,所以推荐使用miniconda。
下载网址
miniconda官网:https://conda.io/miniconda.html
选择适合自己的版本,用wget命令下载。
这里选择的是latest-Linux版本,所以下载的程序会随着python的版本更新而更新(现在下载的版本默认的python版本已经是3.7了)
安装命令
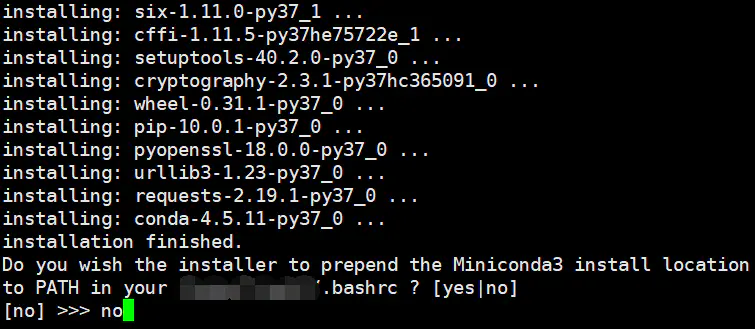
注意,以前的教程都是教一路yes下来的,但是会有隐患,特别是当你的服务器之前有安装过软件的话,conda会污染你原来的环境,把你原来设置好的东西进行更改。具体的惨痛教训请参见:
Anaconda is a snake.
2020-06-14 update: 从下面不加入环境变量这一步开始,其实是非必须的(也就是正常的加入环境变量即可)。之前所谓的会污染环境等等问题可能都是将大量的软件直接安装在conda的base环境中引起的,只要养成好的使用习惯,灵活使用conda create 命令将不同的软件安装到自己单独的虚拟环境中就可以了。把conda这条蟒蛇关进一个一个的笼子里,才能更好的为我们的科研服务~
所以在询问是否将conda加入环境变量的时候选择no。
启动conda(非必须步骤)
2020-06-14 update: 只要正常的按照安装提示进行启动即可
在上一步选择no之后,输入conda是会报找不到此命令的。那要如何启动呢?
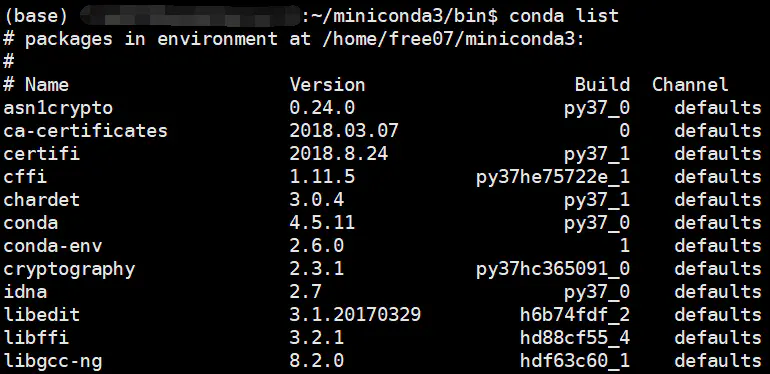
找到你刚才安装的miniconda,如果没有更改过安装位置的话应该是在/home下面,cd到miniconda3的bin目录下面,能看到有一个activate。
这里需要给
activate添加一下权限才能使用接下来启动conda
当命令行前面出现
(base)的时候说明现在已经在conda的环境中了。这时候输入conda list 命令就有反应了添加频道
2019-05-16 update: 这个道理跟家里的电视机是一样一样的,安装conda就相当于买了一台电视机,但是有电视了不意味着你就能看节目了,你要手动添加频道才能看你想看的电视节目。
官方channel: (先不要急着添加这两个哦~,只要添加下面的清华的4个镜像地址就足够了的~):
官方的话这两个channel应该就够了的。
2020-06-14 update:但是其实现在用国内的镜像比较多,官方的频道相较而言速度较慢。但也不是绝对的,有小伙伴跟我说他使用官方的频道也很流畅,所以见仁见智啦。另外,不建议加入大量的相同的频道,如添加了官方的bioconda之后又添加清华的bioconda镜像,没有必要,而且会拖慢速度。
2019-06-12 update:最近在装raxml-ng的时候发现了一个新的channel叫genomedk,各位也可以添加到condarc里去 ?
顺便安利一个我hoptop学长的教程:如何搭建一个本地的conda镜像(包含bioconda)供各位爱折腾的高端玩家。2019-06-17 update: 清华恢复了conda 的镜像了!详情请看下面:
Anaconda 镜像即将恢复
借花献佛给大家复习一遍如何添加清华的镜像channels: 来源 → 生信媛: 喜大普奔: Anaconda的清华镜像又可以用了
2020-06-14 update: 为了分担清华源镜像的压力,最近北京外国语大学也开启了镜像站点,同样是由清华TUNA团队维护的,如果有小伙伴遇到清华源速度很慢的情况的话,可以考虑换成北外的镜像。新闻传送门:https://mirrors.tuna.tsinghua.edu.cn/news/bfsu-mirror/镜像传送门:https://mirrors.bfsu.edu.cn/help/anaconda/2020-08-05 update: 为了方便大家(当然主要是自己偷懒用), 把北外的链接也给写出来, 这样就可以直接复制粘贴了~当然两者取其一就可以了, 不用重复添加.另外, 我查看了中科大的镜像https://mirrors.ustc.edu.cn/anaconda, 点击这个地址会直接跳转到清华tuna的镜像站点. 所以目前看起来国内是只有清华和北外两个镜像站点可用了~如果有小伙伴知道还有别的镜像可以用的话欢迎在下面留言或者私信我鸭!
如果你需要其他的更多的频道,请访问这个地址?: https://mirrors.bfsu.edu.cn/help/anaconda/ 里面有详细的指导哒
2020-08-10 update: 在生信技能树的群里由群友@合肥-生信-gzcdo 提供了两个新的conda的国内镜像源
https://mirrors.nju.edu.cn/anaconda/
https://mirrors.sjtug.sjtu.edu.cn/anaconda/
各位朋友也可以试试看这两个镜像呀!~
显示安装的频道
查看已经添加的channels
已添加的channel在哪里查看
利用conda安装生物信息软件
安装命令:
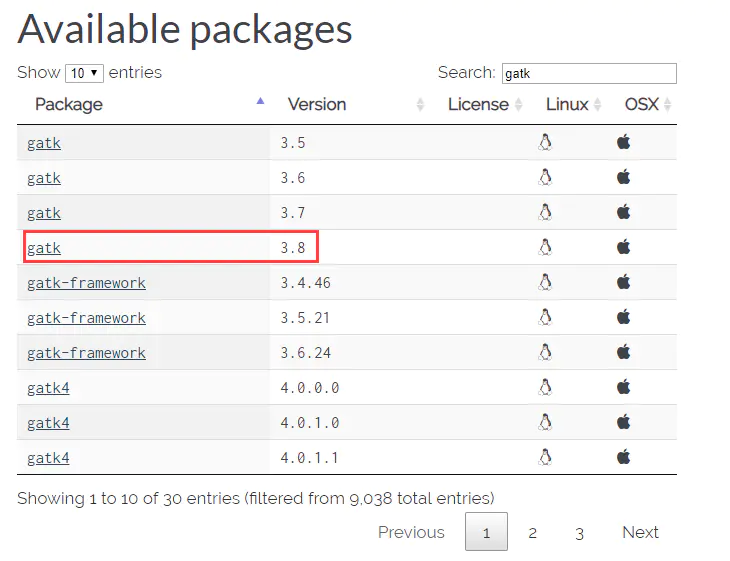
搜索需要的安装包:
提供一个网址,用于事先查找想安装的软件存不存在conda available packages2020-06-14 update: 链接已挂,请选择用下面的conda search命令或者开头提供的更新的网址
当然, 也可以用这个命令进行搜索(会稍微慢一点)
安装完成后,可以用“which 软件名”来查看该软件安装的位置:
如需要安装特定的版本:
这时conda会先卸载已安装版本,然后重新安装指定版本。
查看已安装软件:
更新指定软件:
卸载指定软件:
退出conda环境
退出也很简单,之前我们是. ./activate 或者 (. ~/miniconda3/bin/activate)现在退出只要:
就退出当前的环境了
创建软件的软链接(非必须步骤)
跟着命令一路敲到这里的小旁友们估计发现了,现在退出conda环境之后之前安装的软件全都GG了,敲命令没法执行了!
怎么办呢!其实只要把安装好的软件软连接到一个处在环境变量里的位置就可以使用了。三步走:
- 第一步,创建一个文件夹
我一般的习惯是在/home目录下创建一个.soft文件夹 - 第二步,将这个文件夹添加到环境变量中
- 第三步,软链接
这样就可以运行啦~如果还是不行建议试试初始化一下bashrc:. ./bashrc
创建conda环境(常用步骤,强烈推荐)
之前创建的时候显示的是(base)这是conda的基本环境,有些软件依赖的是python2的版本,当你还是使用你的base的时候你的base里的python会被自动降级,有可能会引发别的软件的报错,所以,可以给一些特别的软件一些特别的关照,比如创建一个单独的环境。
在conda环境下,输入conda env list(或者输入conda info --envs也是一样滴)查看当前存在的环境:
目前只有一个base
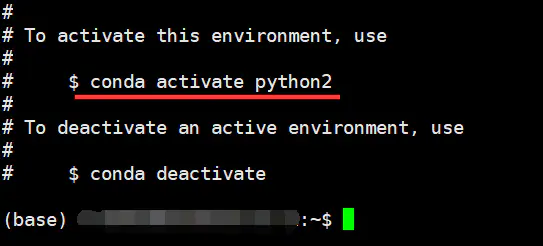
conda会创建一个新的python2的环境,并且会很温馨的提示你只要输入conda activate python2就可以启动这个环境了
退出环境
如上面的截图提到的,只要
2019-6-28 update: 如何删除和重命名一个已存在的环境
删除环境
删除也很容易的
就可以退出当前环境。
掌握了创建和删除我们就可以实现重命名的操作了
重命名环境
实际上conda并没有提供这样的功能,但是可以曲线救国,原理是先克隆一个原来的环境,命名成想要的名字,再把原来的环境删掉即可
参考自:conda 创建/删除/重命名 环境
接下来演示把一个原来叫做py2的环境重新命名成python2:
骚操作:allias简化启动(非必须步骤)
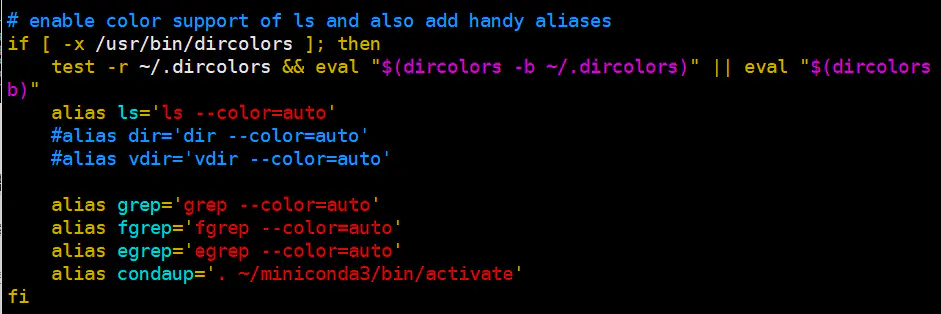
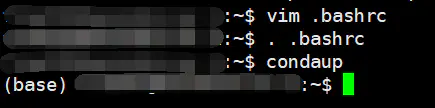
linux提供了一个给大家偷懒的命令叫alias,只要在你的
.bashrc里设置一下就好了,我添加了一条叫做condaup的命令,这样就可以免去每次敲. ~/miniconda/bin/dactivate的麻烦,一步搞定~技术宅改变世界!报错信息集锦
2020-08-06 update: 我最近用conda总是发生一些奇怪的问题, 所以想把报错的信息及解决方式给收集整理一下.
报错1: 网络错误
一般这种问题就只要重新运行一下上一条命令就可以了. 有的时候网络不稳定而已.
报错2: 其他的错误等我找到解决方法再更新上来~
参考及感谢
感谢青山屋主在知乎的专栏文章~
我为什么能知道这么多呢?因为我看了我洲更学长hoptop的课啊!推荐给你们!
https://ke.qq.com/course/3108382020-07-08 update: 洲更学长的课程免费在b站放送啦!~快来白嫖!~
https://www.bilibili.com/video/BV1JJ411p7fXconda的cheatsheet。conda官方提供的小抄了解一下~
https://conda.io/docs/user-guide/cheatsheet.html
ps:简书终于提供markdown编辑器的同步滚动了!开心!






.png)

发表评论 取消回复